ПОЛУЧЕНИЕ ДЕЛЕЦИОННЫХ МУТАНТОВ BACILLUS PUMILUS ПО ГЕНАМ ПРОТЕИНАЗ НА ОСНОВЕ ТЕХНОЛОГИИ CRISPR-CAS9
ПОЛУЧЕНИЕ ДЕЛЕЦИОННЫХ МУТАНТОВ BACILLUS PUMILUS ПО ГЕНАМ ПРОТЕИНАЗ НА ОСНОВЕ ТЕХНОЛОГИИ CRISPR-CAS9
Научная статья
Тойменцева А.А.1, *, Альтенбухнер Дж.2
1 ORCID: 0000-0002-7397-9585, кандидат биологических наук,
Казанский (Приволжский) федеральный университет, Казань, Россия;
2 Штутгартский университет, Штутгарт, Германия
* Корреспондирующий автор (tojmencevaaa[at]mail.ru)
Аннотация
Впервые, с применением технологии CRIPSR-Cas9 редактирования геномов, получен делеционный мутант бактерий Bacillus pumilus с инактивированным геном субтилизиноподобной протеиназы (aprBp). В ходе работы разработан вектор (pToy1.4) для инактивации гена aprBp. Показано, что эффективность трансформации полученной плазмиды в клетки B. pumilus очень низкая, что может быть связано с наличием фагов в геноме этой бактерии, отсутствием генов компетентности, а также большой молекулярной массой разработанного вектора. Штамм с инактивированным геном aprBp интересен для изучения фундаментальной роли этого фермента в клетках B. pumilus, а также для получения гомо- и гетерологичных белков.
Ключевые слова: Bacillus pumilus, протеиназы, редактирование генома, CRIPSR-Cas9, плазмида.OBTAINING BACILLUS PUMILUS DELETION MUTANTS FOR PROTEINASE GENES BASED ON CRISPR-CAS9 TECHNOLOGY
Research article
Toimentseva A.A.1, *, Altenbuchner Dzh.2
1 ORCID: 0000-0002-7397-9585,
Kazan (Volga Region) Federal University, Kazan, Russia;
2 University of Stuttgart, Stuttgart, Germany
* Corresponding author (tojmencevaaa[at]mail.ru)
AbstractDeletion mutant of Bacillus pumilus bacteria with inactivated subtilisin-like proteinase (aprBp) gene was obtained with the help of CRIPSR-Cas9 genome editing technology for the first time. In the course of the work, vector (pToy1.4) was developed for the inactivation of the aprBp gene. It is shown that the efficiency of transformation of the obtained plasmid into B. pumilus cells is very low, which may be due to the presence of phages in the genome of this bacterium, the lack of competence genes, as well as large molecular weight of the developed vector. The strain with the inactivated aprBp gene is of interest for further study because of the fundamental role of this enzyme in B. pumilus cells, as well as in the production of homo- and heterologous proteins.
Keywords: Bacillus pumilus, proteinase, genome editing, CRISPR-Cas9, plasmid.CRISPR-Cas9 (clustered regularly interspaced short palindromic repeats, CRISPR associated protein 9) технология редактирования генома представляет собой современный метод внесения изменений в геном бактерий, растений, различных клеточных линий, отдельных клеток и тканей. С 2012 года была разработана базовая CRISPR-Cas9 технология на основе бактерий Streptococcus pyogenes, которая получила широкое распространение [1], [2]. Система состоит из Cas9 белка, обладающего нуклеазной активностью и двух некодирующих РНК (CRISPR-RNA/crRNA и трансактивирующей CRISPR-RNA/tracrRNA) которые обеспечивают направленную активность Cas9 белка по отношению к определенному участку ДНК. Для удобства работы обе РНК молекулы объединяют в общую молекулу (synthetic single-guide RNA/sgRNA). Таким образом, комплекс Cas9-sgRNA, содержащий 17-30 п.н. гомологичный участок способен узнавать специфический участок в геноме и вносить двухцепочечный разрыв в ДНК. Другим важным компонентом CRISPR-Cas9 системы редактирования является наличие короткой консервативной последовательности, называемой PAM (protospacer adjacent motif), которая расположена рядом с целевым фрагментом генома. Для Cas9-нуклеазы S. pyogenes PAM последовательность представляет собой NNG. Преимуществом использования CRISPR-Cas9 технологии является возможность использования нескольких sgRNA молекул, которые направляют Cas9-нуклеазу сразу к нескольким участкам генома.
Двухцепопчные разрывы в ДНК, образованные CRISPR-Cas9 могут быть восстановлены с помощью двух механизмов – негомологичного восстановления концов ДНК (non-homologous end joining, NHEJ) и гомологичной репарации (homologous recombination, HR). Негомологичное восстановление часто приводит к образованию делеций или инсерций (insertion or deletion/Indels) в целевом участке ДНК. Гомологичная рекомбинация напротив, приводит к точному восстановлению ДНК относительно гомологичной последовательности.
Cas9-нуклеаза S. pyogenes имеет в своем составе два нуклеазных домена, NHN и RuvC-подобный, каждый из которых способен вносить одноцепопчечный разрыв в целевую ДНК, при условии, что Cas9-нуклеаза связана с sgRNA. Если один из доменов нуклеазы несет в себе мутацию, Cas9-sgRNA функционирует как никаза (надрезающая эндонуклеаза, которая режет лишь одну цепь двухцепочечной ДНК в определенных последовательностях нуклеотидов). Когда мутации содержат оба нуклеазных домена, Cas9-нуклеаза не обладает никакой активностью, но способна функционировать как программируемый ДНК-связывающий белок (dead Cas9/dCas9). На основе описанных модификаций созданы наборы плазмид, позволяющие добиваться различных модификаций в геноме.
В настоящей работе CRISPR-Cas9 технология была использована для редактирования генома бактерий Bacillus pumilus. Бактерии B. pumilus известны своей способностью к продукции (секреции) гидролитических ферментов [3], [4]. Протеазы широко применяются в различных отраслях – фармацевтике и медицине, сельском хозяйстве, пищевой и текстильной индустриях [5]. Для направленного получения целевого фермента часто применяют штаммы бактерий с делетированными генами протеиназ [6], [7], [8], т.е. добиваются условий, когда экспрессия генов протеиназ не влияет на продукцию целевых ферментов. Кроме того, получение таких делеционных мутантов позволяет понять фундаментальную роль отдельных протеиназ для бактериальной клетки.
Материалы и методы
В работе использовали штаммы B. pumilus 3-19 (устойчивый к стрептомицину), Escherichia coli JM109 и плазмиду pJOE9282.1. Генотип штаммов использованных и полученных в работе представлен в таблице 1.
В качестве питательных сред использовали LB-агар, LB-бульон, минимальную питательную среду (МС) Spizizen (при необходимости с добавлением 2% агара) [9]. Антибиотики добавляли в среду в концентрациях 10 мкг/мл (канамицин), 100 мкг/мл (ампициллин), 500 мкг/мл (стрептомицин). Ксилозу добавляли в LB-агар к концентрации 0,2 %.
Выделение плазмидной ДНК из клеток E. coli проводили щелочным методом [10]. Рестрикцию, лигирование ДНК и ПЦР осуществляли в соответствии с рекомендациями фирм-изготовителей ферментов (Fermentas, New England BioLab). Гибридизацию праймеров (s12122/12123) проводили в объёме 10 мкл по программе: 1 мин - 95ºC; 10 мин - 65ºC; остывание при комнатной температуре. Концентрация праймеров для гибридизации составляла 100 пМоль/мкл. ПЦР для получения фланкирующих последовательностей гена aprBp с геномной ДНК штамма B. pumilus 3-19 проводили с использованием Phusion полимеразы (Phusion High-Fidelity DNA Polymerase, New England Biolabs, #M0530S). Электрофорез ДНК проводили в 1% агарозном геле в трис-ацетатном буфере. В качестве маркеров использовали 1 Кб набор фирмы Fermentas (кат. номер #0333). Трансформацию рекомбинантных ДНК в клетки E. coli проводили с использованием CaCl2 [10]. Определение нуклеотидной последовательности ДНК проводили по методу Сэнгера в компании GATC Biotech (Германия). Для обработки результатов секвенирования использовали программу Clone Manager 9.
Таблица 1 – Использованные в работе штаммы бактерий
| Название штамма | Генотип | Цель использования | Источник |
| B. pumilus 3-19 | strR | Делеция генов протеиназ, штамм-реципиент | Коллекция лаборатории (кафедра микробиологии ИФМиБ, НИЛ биосинтеза и биоинженерии ферментов, КФУ) |
| E. coli JM109 | endA1, glnV44, thi-1, relA1, gyrA96, recA1, mcrB+, Δ(lac-proAB), e14-[F' traD36 proAB+ lacIq lacZΔM15], hsdR17(rK-mK+) | Получение рекомбинантных плазмид | Коллекция лаборатории (кафедра микробиологии ИФМиБ, НИЛ биосинтеза и биоинженерии ферментов, КФУ) |
| B. pumilus 023 | strR, ΔaprBp | Штамм с инактивированным геном субтилизиноподобной протеиназы | Получен в работе |
Результаты и обсуждение
Нами использован шаттл-вектор pJOE9282.1 на основе системы CRISPR-Cas9 II типа S. pyogenes [11]. В состав pJOE9282.1 плазмиды входят температура-чувствительный ориджин репликации pE194ts для клеток бацилл, ориджин репликации pUC для бактерий E. coli, ген устойчивости к канамицину экспрессирующийся как в клетках E. coli так и в клетках бацилл, cas9 ген под контролем ксилоза-индуцируемого промотора (Pxyl) и некодирующая РНК (sgRNA). Карта pJOE9282.1 плазмиды представлена на рисунке 1.
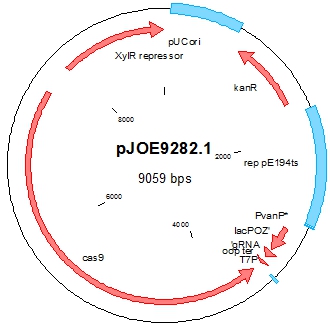
Рис. 1 – Генетическая карта плазмиды pJOE9282.1
Для редактирования генома B. pumilus ген сериновой протеиназы aprBp был выбран в качестве мишени для инактивации. Сериновая протеиназа AprBp является «мажорным» секретируемым ферментом протеолитического пула клеток B. pumilus. До 70% протеолитической активности в клетках B. pumilus приходится на AprBp фермент [12]. Однако, функция данного фермента в клетках бацилл остается до конца не изученной, поэтому получение штамма с инактивированным aprBp геном является важной фундаментальной задачей. Для делеции aprBp гена в составе хромосомы бактерий B. pumilus проводили следующие процедуры. Плазмида pJOE9282.1 для инактивации гена aprBp была предоставлена доктором Дж. Альтенбухнером, эта плазмида является производной от плазмиды pJOE8999 [11]. Плазмиду pJOE9282.1 расщепляли по сайту рестрикции BsaI и проводили интеграцию спейсерного фрагмента, полученного путём гибридизации праймеров s12122/12123. Далее, два ПЦР фрагмента (`aprBp-L (765 п.о.) и aprBp`-R (533п.о.)), полученных с геномной ДНК B. pumilus, клонировали по сайту SfiI. Использованные праймеры представлены в таблице 2.
Таблица 2 – Олигонулеотиды использованные в работе
| Название | Последовательность 5’→3’* |
| спейсерный фрагмент** | |
| s12122 | tacgTTCGGACATCTTTACTTGTG |
| s12123 | aaacCACAAGTAAAGATGTCCGAA |
| `aprBp-L*** | |
| s12124 | aaGGCCaacgaGGCCAACAAGTCCTGATCGTGGCA |
| s12125 | aaGGCCatgttGGCCGCCATTTTGAGTGACGGCTT |
| aprBp`-R*** | |
| s12126 | aaGGCCaacatGGCCATAACCGCGGAGTCGTTGTT |
| s12127 | aaGGCCttattGGCCTTCCGCTATGGACACCCAAC |
| cas9 | |
| s12092 | CGCGTGGCAATAGTCGTTTT |
| s12093 | ATGCCGCCCCATTACTTTGA |
| yrhJ | |
| s11949 | GGAGGCTAGCAGATGAATTAGG |
| s11950 | CGAAACAACAAGCGGGC |
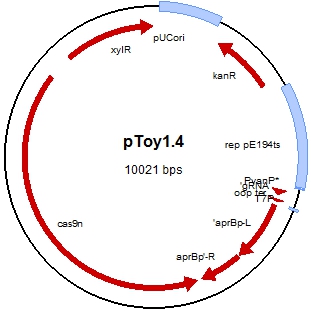
Рис. 2 – Генетическая карта плазмиды pToy1.4
Проводили секвенирование pToy1.4 плазмиды. В результате секвенирования были подтверждены спейсерная нуклеотидная последовательность и последовательности `aprBp-L и aprBp`-R фрагментов в составе плазмиды pToy1.4. Таким образом, нами сконструирована плазмида для инактивации гена aprBp протеиназы в геноме B. pumilus.
Интеграцию плазмиды pToy1.4 (в концентрации 1 мкг) в клетки бацилл выполняли методом транформации с использованием минимальных сред. Клетки B. pumilus после трансформации инкубировали на питательной среде со стрептомицином и канамицином при температуре +30ºC в течение двух дней. Эффективность трансформации pToy1.4 плазмиды в клетки B. pumilus была низкой – все 8 колоний были отобраны для дальнейшего анализа.
Подтверждение трансформации pToy1.4 плазмиды проводили с помощью ПЦР с колоний с использованием пар праймеров s12092/s12093 к гену cas9, а также пар праймеров s11949/s11950 к yrhJ гену (кодирующему редуктазу/NADPH-hemoprotein reductase) с хромосомной ДНК B. pumilus. Результаты ПЦР представлены на рисунке 7 и 8.
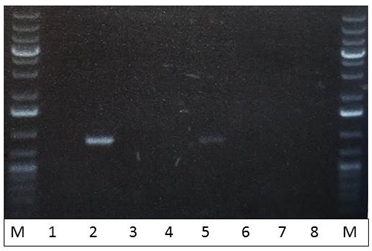
Рис. 3 – ПЦР с использованием пар праймеров s12092/s12093 к гену cas9: молекулярная масса ПЦР продукта с применением этих праймеров – 896 п.о. 1-8: колонии B. pumilus, полученные после трансформации pToy1.4 плазмиды; М – маркер молекулярного веса #0333 (Fermentas)
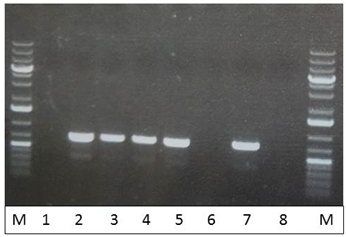
Рис. 4 – ПЦР с использованием пар праймеров s11949/s11950 к yrhJ гену B. Pumilus: молекулярная масса ПЦР продукта с применением этих праймеров – 726 п.о. 1-8: колонии B. pumilus, полученные после трансформации pToy1.4 плазмиды; М – маркер молекулярного веса #0333 (Fermentas)
Как видно из рисунка 3 – только две колонии (№2 и №5) содержали в себе плазмиду pToy1.4. Результаты ПЦР с использованием праймеров s11949/s11950 показали, что пять колоний являются представителями B. pumilus. Таким образом, нами получены 2 колонии B. pumilus (№2 и №5), которые содержат в своем составе плазмиду pToy1.4 для инактивации гена aprBp и являются представителями вида B. pumilus.
Далее нами проведены процедуры для инактивации гена aprBp. Т.к. ген cas9 нуклеазы находится под контролем ксилоза-индуцируемого промотора, отобранные колонии (№2 и №5) культивировали в течение ночи на питательной среде LB-агар при +30ºC, содержащей ксилозу. Клетки пересевали на LB-агар до получения отдельных колоний и выращивали при +42ºC для удаления плазмиды (т.к. плазмида pToy1.4 имеет температура-чувствительный ориджин репликации pE194ts - повышение температуры должно приводить к потере плазмиды). С полученных колоний проводили ПЦР с использованием праймеров s12124/s12127. Результаты ПЦР представлены на рисунках 5 и 6.
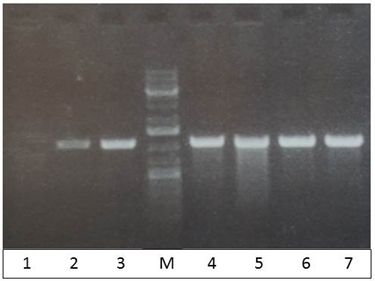
Рис. 5 – ПЦР с колоний B. pumilus (исходная колония №2 после трансформации, активации гена cas9 и удаления pToy1.4. плазмиды) с использованием пар праймеров s12124/s12127: 1 – отрицательный контроль (H2O); 2 – положительный контроль с плазмиды pToy1.4; 3-7 – ПЦР с колоний; М – маркер молекулярного веса #0333 (Fermentas)
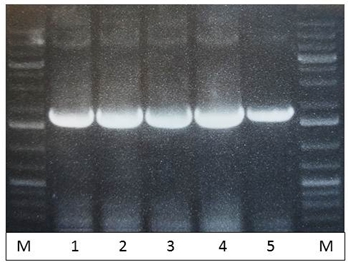
Рис. 6 – ПЦР с колоний B. pumilus (исходная колония №5 после трансформации, активации гена cas9 и удаления pToy1.4. плазмиды) с использованием пар праймеров s12124/s12127: 1-5 – ПЦР с колоний; М – маркер молекулярного веса #0333 (Fermentas)
Эти результаты свидетельствуют о том, что из двух полученных трансформантов только одна колония (№2) содержала плазмиду pToy1.4 и привела к успешному удалению гена субтилизиноподобной протеиназы в геноме штамма B. pumilus 3-19. Разработанному в ходе работы штамму (с генотипом strR, ΔaprBp) присвоен номер B. pumilus 023.
Таким образом, впервые, с помощью применения технологии CRISPR-Cas9, нам удалось редактировать геном бактерий вида B. pumilus. Необходимо отметить, что эффективность трансформации плазмиды содержащей ген cas9 S. pyogenes была очень низкой. Известно, что многие штаммы вида B. pumilus обладают низкой природной компетентностью (способностью принимать чужеродную ДНК). Одной из причин этого может являться наличие в геноме фагов, например phi105 фага [13] [14]. Трудности с трансформацией ДНК могут быть связаны также с большим молекулярным весом плазмиды (для плазмиды pToy1.4 молекулярная масса составляет больше 10 Кб), отсутствием генов компетентности (например, оперона comKcomS). Полученный в ходе работы штамм B. pumilus 023 будет использован для описания морфологических и физиологических особенностей, а также будет выполнен анализ протеомного профиля данного делеционного мутанта.
| Финансирование Работа выполнена в рамках государственной программы повышения конкурентоспособности Казанского (Приволжского) федерального университета среди ведущих мировых научно-образовательных центров и при финансовой поддержке РФФИ в рамках научного проекта № 16-34-60198 мол_а_дк. | Funding The work was performed as part of the state program to improve the competitiveness of the Kazan (Volga Region) Federal University among the world's leading research and education centers and with financial support from the Russian Foundation for Basic Research within the framework of research project No.16-34-60198 mol_a_dk. |
| Конфликт интересов Не указан. | Conflict of Interest None declared. |
Список литературы / References
- Deltcheva E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III / E. Deltcheva, K. Chylinski, C. M. Sharma and others // Nature. – 2011. - Vol. 471(7340). - P. 602-7. doi: 10.1038/nature09886
- Jinek M. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity / M. Jinek, K. Chylinski, I. Fonfara and others // Science. – 2012. – Vol. 337(6096). – P. 816-21. doi: 10.1126/science.1225829
- Sharipova M. R. The expression of Bacillus intermedius glutamyl endopeptidase gene in Bacillus subtilis recombinant strains / M. R. Sharipova, E. I. Shagimardanova, I. B. Chastukhina и др. // Mol Biol Rep. – 2007. – Vol. 34(2). – P. 79-87.
- Sharipova M. The expression of the serine proteinase gene of intermedius in B. subtilis / M. Sharipova, N. Balaban, A. Kayumov // Microbiological Research. – 2008. – Vol. 163(1). – P. 39-50.
- Contesini F. J. An overview of Bacillus proteases: from production to application / F. J. Contesini, R. R. Melo, H. H. Sato // Critical Reviews in Biotechnology. – 2018. – Vol. 38(3). – P. 321-334. doi: 10.1080/07388551.2017.1354354
- Wu X. C. Engineering a Bacillus subtilis expression-secretion system with a strain deficient in six extracellular proteases / X. C. Wu, W. Lee, L. Tran and others // J. Bacteriol. – 1991. – Vol. 173(16). – P. 4952–4958.
- Westers Bacillus subtilis as cell factory for pharmaceutical proteins: a biotechnological approach to optimize the host organism / L. Westers, H. Westers, W. J. Quax // Biochim. Biophys. Acta. – 2004. – Vol. 1694(1-3). – P. 299-310. doi: 10.1016/j.bbamcr.2004.02.011.
- Li Y. Characterization of genome-reduced Bacillus subtilis strains and their application for the production of guanosine and thymidine / Y. Li, X. Zhu, X. Zhang and others // Microbial Cell Factories. – 2016. – Vol. 15(94). doi.org/10.1186/s12934-016-0494-7
- Harwood C. R. Molecular biological methods for Bacillus / C. R. Harwood, S. M. Cutting // Chichester. New York: Wiley. – 1990. – P. XXXV, 581.
- Das S. Microbial biotechnology - a laboratory manual for bacterial systems / S. Das, H. R. Dash. – Switzerland: Springer, 2015. – P. 239. doi: 10.1007/978-81-322-2095-4_2.
- Altenbuchner J. Editing of the Bacillus subtilis Genome by the CRISPR-Cas9 System / J. Altenbuchner // Appl. Environ. Microbiol. - 2016. –V. 82(17). –P. 5421-5427. doi:1128/AEM.01453-16
- Кириллова Ю. М. Особенности биосинтеза субтилизиноподобной протеиназы Bacillus intermedius рекомбинантным штаммом B. subtilis / Ю. М. Кириллова, Е. О. Михайлова, Н. П. Балабан и др. // Микробиология. - 2006. -Т. 75(2) - С. 179-185.
- Garro A. J. Relationship between lysogeny, spontaneous induction, and transformation efficiencies in Bacillus subtilis / A. J. Garo, M. F. Law // J. Bacteriol. – 1974. – Vol. 120. – P. 1256-1259.
- Bose B. A conserved anti-repressor controls horizontal gene transfer by proteolysis / B. Bose, J. M. Auchtung, C. A. Lee and others // Mol. Microbiol. – 2008. – Vol. 70. – P. 570-582. doi: 10.1111/j.1365-2958.2008.06414.x.
Список литературы на английском языке / References in English
- Deltcheva E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III / E. Deltcheva, K. Chylinski, C. M. Sharma and others // Nature. – 2011. - Vol. 471(7340). - P. 602-7. doi: 10.1038/nature09886
- Jinek M. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity / M. Jinek, K. Chylinski, I. Fonfara and others // Science. – 2012. – Vol. 337(6096). – P. 816-21. doi: 10.1126/science.1225829
- Sharipova M. R. The expression of Bacillus intermedius glutamyl endopeptidase gene in Bacillus subtilis recombinant strains / M. R. Sharipova, E. I. Shagimardanova, I. B. Chastukhina и др. // Mol Biol Rep. – 2007. – Vol. 34(2). – P. 79-87.
- Sharipova M. The expression of the serine proteinase gene of intermedius in B. subtilis / M. Sharipova, N. Balaban, A. Kayumov // Microbiological Research. – 2008. – Vol. 163(1). – P. 39-50.
- Contesini F. J. An overview of Bacillus proteases: from production to application / F. J. Contesini, R. R. Melo, H. H. Sato // Critical Reviews in Biotechnology. – 2018. – Vol. 38(3). – P. 321-334. doi: 10.1080/07388551.2017.1354354
- Wu X. C. Engineering a Bacillus subtilis expression-secretion system with a strain deficient in six extracellular proteases / X. C. Wu, W. Lee, L. Tran and others // J. Bacteriol. – 1991. – Vol. 173(16). – P. 4952–4958.
- Westers Bacillus subtilis as cell factory for pharmaceutical proteins: a biotechnological approach to optimize the host organism / L. Westers, H. Westers, W. J. Quax // Biochim. Biophys. Acta. – 2004. – Vol. 1694(1-3). – P. 299-310. doi: 10.1016/j.bbamcr.2004.02.011.
- Li Y. Characterization of genome-reduced Bacillus subtilis strains and their application for the production of guanosine and thymidine / Y. Li, X. Zhu, X. Zhang and others // Microbial Cell Factories. – 2016. – Vol. 15(94). doi.org/10.1186/s12934-016-0494-7
- Harwood C. R. Molecular biological methods for Bacillus / C. R. Harwood, S. M. Cutting // Chichester. New York: Wiley. – 1990. – P. XXXV, 581.
- Das S. Microbial biotechnology - a laboratory manual for bacterial systems / S. Das, H. R. Dash. – Switzerland: Springer, 2015. – P. 239. doi: 10.1007/978-81-322-2095-4_2.
- Altenbuchner J. Editing of the Bacillus subtilis Genome by the CRISPR-Cas9 System / J. Altenbuchner // Appl. Environ. Microbiol. - 2016. –V. 82(17). –P. 5421-5427. doi:1128/AEM.01453-16
- Kirillova YU. M. Osobennosti biosinteza subtilizinopodobnoj proteinazy Bacillus intermedius rekombinantnym shtammom B. subtilis [Biosynthesis of the Bacillus intermedius subtilisin-like serine proteinase by the recombinant Bacillus subtilis strain B. subtilis] / YU. M. Kirillova, E. O. Mihajlova, N. P. Balaban i dr. // Mikrobiologiya [Microbiology]. - 2006. -Vol. 75(2) - P. 179-185.Garro A. J. Relationship between lysogeny, spontaneous induction, and transformation efficiencies in Bacillus subtilis / A. J. Garo, M. F. Law // J. Bacteriol. – 1974. – Vol. 120. – P. 1256-1259. [in Russian]
- Bose B. A conserved anti-repressor controls horizontal gene transfer by proteolysis / B. Bose, J. M. Auchtung, C. A. Lee and others // Mol. Microbiol. – 2008. – Vol. 70. – P. 570-582. doi: 10.1111/j.1365-2958.2008.06414.x.
