ОЦЕНКА СОДЕРЖАНИЯ ГЕНОВ АНТИБИОТИКОРЕЗИСТЕНТНОСТИ В ПОЧВАХ Г. РОСТОВА-НА-ДОНУ
ОЦЕНКА СОДЕРЖАНИЯ ГЕНОВ АНТИБИОТИКОРЕЗИСТЕНТНОСТИ В ПОЧВАХ Г. РОСТОВА-НА-ДОНУ
Научная статья
Ажогина Т.Н.1, *, Сазыкина М.А.2
1 ORCID: 0000-0003-0274-783X;
2 ORCID: 0000-0001-6974-3361;
1–2 Южный федеральный университет, Ростов-на-Дону, Россия
* Корреспондирующий автор (t.azhogina[at]mail.ru)
Аннотация
В настоящее время гены резистентности к антибиотикам (АРГ) стали новым видом загрязнения окружающей среды. Но информации, касающейся диссеминации АРГ в природных экосистемах, крайне мало. В данной работе исследовано присутствие 8 АРГ (sulII, TetO, aadA2, VanB, ermB, mphA, VIM, MecA) в почвах, отобранных на территории г. Ростована-Дону в 2020 и 2022 гг. Методом ПЦР в реальном времени в большинстве почв были обнаружены все исследованные АРГ, кроме MecA. Ген MecA был обнаружен только в одном образце почв, отобранном в 2020 году. Наиболее часто встречался ген устойчивости к сульфаниламидам (sulII). Количество копий генов VanB, TetO, ErmB, mphA, VIM выросло, а генов sulII и aadA2 снизилось в 2022 году относительно количества копий АРГ 2020 года.
Ключевые слова: гены резистентности к антибиотикам, почвы, мониторинг, загрязнение.
ASSESSMENT OF ANTIBIOTIC RESISTANCE GENES CONTENT IN SOILS OF ROSTOV-ON-DON
Research article
Azhogina T.N.1, *, Sazykina M.A.2
1 ORCID: 0000-0003-0274-783X;
2 ORCID: 0000-0001-6974-3361;
1–2 Southern Federal University, Rostov-on-Don, Russia
* Corresponding author (t.azhogina[at]mail.ru)
Abstract
Today, antibiotic resistance genes (ARG) have become a new type of environmental pollution. However, there is very little information on the dissemination of ARG in natural ecosystems. In this paper, the presence of 8 ARG (sulII, TetO, aadA2, VanB, ermB, mphA, VIM, MecA) in soils sampled in Rostov-on-Don in 2020 and 2022 was examined. By real-time PCR method, all the studied ARG were detected in most soils, except for MecA. The MecA gene was found in only one soil sample taken in 2020. The most common was the sulfanilamide resistance gene (sulII). The number of copies of the vanB, TetO, ermB, mphA, VIM genes increased, and the sulII and aadA2 genes decreased in 2022 relative to the number of copies of the ARG in 2020.
Keywords: antibiotic resistance genes, soils, monitoring, pollution.
Введение
Появление антибиотиков в 20 веке произвело революцию в современной медицине и животноводстве. Однако устойчивое селекционное давление антибиотиков на бактерии из-за их неправильного и чрезмерного использования во всем мире, привело к увеличению распространения генов резистентности к антибиотикам (АРГ) у широкого спектра клинических патогенов и комменсальных бактерий, и, следовательно, к появлению антибиотикоустойчивых патогенов и «супербактерий» [14], [15].
Чтобы полностью понять механизм возникновения лекарственной устойчивости, нужно обратиться к изучению генов устойчивости к антибиотикам не только в клиниках, но и в естественной природной среде их обитания. Окружающая среда представляет собой важный резервуар и путь передачи устойчивости к противомикробным препаратам [2], [12]. Потенциальный перенос АРГ от бактерий из окружающей среды к патогенам человека может серьезно снизить эффективность антибактериальной терапии, тем самым создавая опасный риск для здоровья населения [11].
Наиболее богатым резервуаром АРГ, благодаря разнообразным микробным сообществам, является почва [6], [7]. Имеются сведения о значительной положительной корреляции между АРГ и антибиотиками в почвенной среде [8], [5]. Чрезмерное использование антибиотиков оказывает избирательное давление на микроорганизмы, что усугубляет загрязнение АРГ [10], [4]. Загрязнение АРГ в почвенной среде достигло больших масштабов, причем содержание некоторых из них выросло более чем в 15 раз с 1970-х годов [9].
Неуклонно растет количество исследований, свидетельствующих о влиянии тяжелых металлов, полихлорированных бифенилов, пестицидов и дезинфицирующих средств на отбор и распространение антибиотикорезистентных бактерий и АРГ [1]. Также хорошо известно, что большое количество органических загрязнителей оказывает значительное давление на селекцию устойчивых к антибиотикам бактерий и АРГ [3].
В связи с этим актуальной задачей является мониторинг содержания генов антибиотикорезистентности в почвах, подверженных значительному антропогенному воздействию. Целью данной работы стал поиск АРГ в подверженных антропогенному прессингу почвах г. Ростова-на-Дону.
Исследования, направленные на экологические проблемы, связанные с развитием резистентности к антибиотикам, помогут найти и внедрить адекватные меры, направленные против распространения бактерий, устойчивых к антибиотикам, в почвах.
Материалы и методы исследования
Для проведения исследования по выделению генов устойчивости к антибиотикам на территории г. Ростова-на-Дону был произведен отбор 14 проб почв (№1–7 рекреационных зон, №8–11 – промышленных зон и №12–14, отобранных на территории больниц) в апреле 2020 и 2022 гг.
Тотальную метагеномную ДНК выделяли из почвы с помощью набора FastDNA для почвы (MPBiomedicals, США). Концентрацию выделенной ДНК измеряли с помощью флуориметра Qubit 3.0 (ThermoFisherScientific, США). Выделенную тотальную ДНК хранили при -20 °C перед последующей количественной ПЦР (qPCR). Определение АРГ проводили с помощью ПЦР в реальном времени.
Были исследованы восемь АРГ, включая гены устойчивости к макролидам (ErmB и mphA), сульфонамидам (sulII), тетрациклину (TetO), карбапенемам (VIM), ванкомицину (VanB), аминогликозидам (aadA2), цефалоспоринам (MecA).
Для нормализации количества АРГ в каждом образце также определяли количество генов 16S рРНК. Для расчета числа копий АРГ и 16S рРНК использовали калибровочную кривую, полученную с использованием 10-кратных серийных разведений (102–108 копий) стандартной плазмиды, содержащей продукт амплификации для каждого праймера. Все q-PCR были выполнены в трех повторностях для каждого образца. Значения эффективности усиления (R2) находились в диапазоне 93–110%. Чтобы свести к минимуму различия, связанные с аналитической эффективностью, дифференциальной экстракцией и вариациями фоновых локальных количеств бактериальных генов, количество АРГ выражалось как отношение количества АРГ к содержанию гена 16S рРНК.
Результаты и обсуждение
На рисунке 1 представлены результаты определения АРГ в почвах г. Ростова-на-Дону.
Как видно представленных на рисунке 1 данных, ген sulIIвстречался в наибольшем количестве практически во всех исследуемых образцах, а ∑АРГ была выше в образцах почв, отобранных в 2022 году.
В образцах почв промышленного назначения зарегистрирована меньшая сумма АРГ. В образцах почв больниц – максимальная. Для почв рекреационных территорий зарегистрирован средний уровень ∑АРГ. Это может быть связано с тем, что микробные сообщества этих территорий не нарушены и в их состав входят бактерии разнообразных групп, которые могут нести различные АРГ.
В образцах № 1-7, № 10 и № 13 наблюдалось увеличение спектра обнаруженных АРГ в 2022 году по сравнению с 2020 годом. В образцах № 9, 11 и 12 напротив, наблюдалось, уменьшение спектра обнаруженных АРГ. В образцах № 8 и 14 количество найденных АРГ не изменилось.
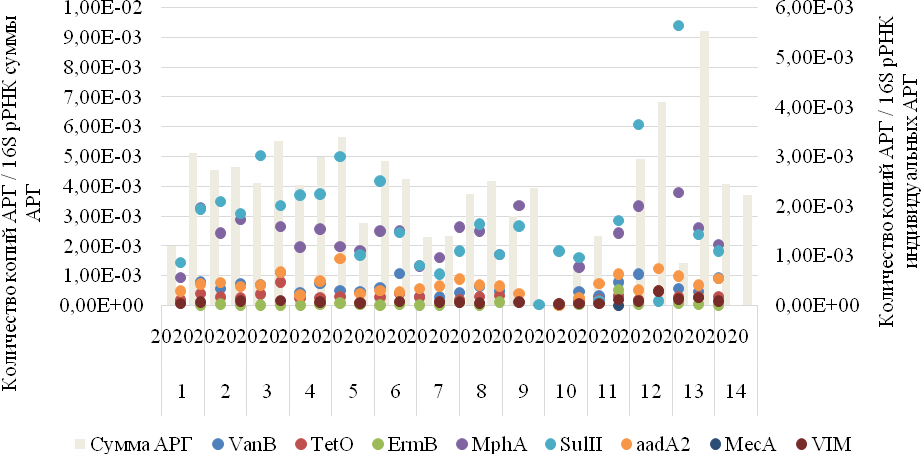
Рис. 1 – Количество копий АРГ / 16S рРНК в исследуемых почвах:
1–7 почвы рекреационных зон, 8–11 – почвы промышленных зон;12–14 почвы на территории больниц)
В 2020 году наибольшая копийность была характерна для гена sulII, затем по убываниюmphA, aadA2, VanB, VIM, TetO, ermB, MecA. В 2022 году – sulII, mphA, aadA2, VanB, TetO, VIM, ermB. Ген MecA в 2022 году обнаружен не был.
В 2020 году все восемь исследуемых генов устойчивости к антибиотикам были обнаружены в образце №12, отобранном на территории больницы. В образце №10, относящемся к промышленным, был обнаружен только один ген – sulII. Максимальная ∑АРГ была обнаружена в образце №5 (5,67×10-3 копий/16S рРНК).
В 2022 году ни в одном из образцов не был обнаружен ген MecA. Семь исследуемых генов были обнаружены в образцах №1-8, 13 и 14. Минимальный спектр (5 АРГ) был обнаружен в образце №10. Наибольшая ∑АРГ была обнаружена в образце №13 (9,21×10-3 копий/16S рРНК).
В целом, в образцах №1-6, 10 и 13 наблюдалось увеличение спектра обнаруженных АРГ в 2022 году относительно спектра АРГ 2020 года. Количество копий генов VanB, TetO, ErmB, mphA, VIM выросло, а генов sulII и aadA2 снизилось в 2022 году относительно количества копий АРГ 2020 года.
В почвах рекреационного назначения наблюдалось увеличение суммы АРГ в 57,2% проб (4 из 7); в почвах промышленного назначения – в 75% (3 из 4); в почвах больниц – в 66,6% (2 из 3). Это может быть связано с растущим антропогенным прессингом, который способствует отбору бактерий, несущих детерминанты резистентности.
Таким образом, полученные данные свидетельствуют о повсеместном распространении исследованных АРГ в почвах г. Ростова-на-Дону. Следует отметить, что городские почвы, в сравнении с другими, менее изучены в плане присутствия и распространенности АРГ (Qiaoetal., 2018; Zhuangetal., 2021). Но почвы городов благоприятны для пролиферации АРГ, поэтому крайне важно постоянно контролировать их распространение.
Заключение
Во всех исследованных почвах г. Ростова-на-Дону обнаружены все изученные АРГ (ErmB, mphA, sulII, TetO, VIM, VanB, aadA2, MecA),кроме MecA. Ген MecA был обнаружен только в одной почве в 2020 году. Наиболее часто встречался ген устойчивости к сульфаниламидам (sulII). Количество копий генов VanB, TetO, ErmB, mphA, VIM выросло, а генов sulII и aadA2 снизилось в 2022 году относительно количества копий АРГ в 2020 году. Полученные результаты свидетельствуют о необходимости постоянного экологического контроля за содержанием и диссеминацией АРГ.
|
Конфликт интересов Не указан. |
Conflict of Interest None declared. |
Список литературы
Azhogina T.N. Influence of pollutants on the spread of antibiotic resistance genes in the environment / T.N. Azhogina, S.G. Skugoreva, A.A.K. Al-Rhammahi et al. // Theoretical and applied ecology. – 2020. – № 3. – P. 6-14.
Berendonk T.U. Tackling antibiotic resistance: the environmental framework / T.U. Berendonk, C.M. Manaia, C. Merlin et al. // Nature reviews microbiology. – 2015. – Vol. 13. – № 5. – P. 310-317.
Chen B. Polycyclic aromatic hydrocarbons (PAHs) enriching antibiotic resistance genes (ARGs) in the soils / B. Chen, R. He, K. Yuan et al. // Environmental pollution. – 2017. – Vol. 220. – P. 1005-1013.
Chen Z. Influence of dissolved organic matter on tetracycline bioavailability to an antibiotic-resistant bacterium / Z. Chen, Y. Zhang, Y. Gao et al. // Environmental science & technology. – 2015. – Vol. 49. – № 18. – P. 10903-10910.
Cheng W. Behavior of antibiotics and antibiotic resistance genes in eco-agricultural system: a case study / W. Cheng, J. Li, Y. Wu et al. // Journal of hazardous materials. – 2016. – Vol. 304. – P. 18-25.
D’Costa V.M. Antibiotic resistance is ancient / V.M. D’Costa, C.E. King, L. Kalan et al. // Nature. – 2011. – Vol. 477. – № 7365. – P. 457-461.
Forsberg K.J. The shared antibiotic resistome of soil bacteria and human pathogens / K.J. Forsberg, A. Reyes, B. Wang et al. // Science. – 2012. – Vol. 337. – №. 6098. – P. 1107-1111.
Huang X. Occurrence and distribution of veterinary antibiotics and tetracycline resistance genes in farmland soils around swine feedlots in Fujian Province, China / X. Huang, C. Liu, K. Li et al. //Environmental Science and Pollution Research. – 2013. – Vol. 20. – №. 12. – P. 9066-9074.
Knapp C.W. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940 / C.W. Knapp, J. Dolfing, P.A. Ehlert et al. // Environmental science & technology. – 2010. – Vol. 44. – № 2. – P. 580-587.
Luo Y.I. Trends in antibiotic resistance genes occurrence in the Haihe River, China / Y.I. Luo, D. Mao, M. Rysz et al. // Environmental science & technology. – 2010. – Vol. 44. – № 19. – P. 7220-7225.
Pehrsson E.C. Interconnected microbiomes and resistomes in low-income human habitats / E.C. Pehrsson, P. Tsukayama, S. Patel et al. //Nature. – 2016. – Vol. 533. – № 7602. – P. 212-216.
Pruden A. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado / A. Pruden, R. Pei, H. Storteboom et al. // Environmental science & technology. – 2006. – Vol. 40. – № 23. – P. 7445-7450.
Qiao M. Review of antibiotic resistance in China and its environment. / M. Qiao, G.G. Ying, A.C. Singer et al. // Environment international. – 2018. – № 110. P. 160-172. DOI: 10.1016/j.envint.2017.10.016
Wenzel R.P. Managing antibiotic resistance / R.P. Wenzel, M.B. Edmond // New England Journal of Medicine. – 2000. – Vol. 343. – № 26. – P. 1961-1963.
Zhuang M. Distribution of antibiotic resistance genes in the environment / M. Zhuang, Y. Achmon, Y. Cao et al. // Environmental Pollution. – 2021. – № 285. – P. 117402. DOI: 10.1016/j.envpol.2021.117402.
