МЕТОД ИЗУЧЕНИЯ НУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ BLV
Смирнов П.Н.1, Батенёва Н.В.2, Князев С.П.3, Хрипко Ю.И.4, Скачков А.С.5
1Доктор ветеринарных наук, 2кандидат биологических наук, 3кандидат биологических наук, 4аспирант, 5аспирант, Новосибирский государственный аграрный университет
МЕТОД ИЗУЧЕНИЯ НУКЛЕОТИДНЫХ ПОСЛЕДОВАТЕЛЬНОСТЕЙ BLV
Аннотация
На протяжении многих лет изучение BLV является актуальной проблемой. Так как до настоящего времени не выявлены многие биологические особенности течения вируса. Мы проанализировали последовательности LTR-области 11 изолятов BLV, в том числе циркулирующих на поголовье скота на территории Российской Федерации. Подобрали наиболее специфичные и чувствительные праймеры, фланкирующие наиболее вариабельный участок LTR-области.
Ключевые слова: вирус лейкемии крупного рогатого скота, генотипирование, ПДРФ-анализ, LTR-область, болезни животных, вирусы, биотехнология животных
Smirnov P.N.1, Bateneva N.V.2, Knyazev S.P.3, Khripko Yo.I.4, Skachkov A.S.5
1 Doctor of veterinary medicine, 2 candidate of biological Sciences, 3 candidate of biological Sciences, 4 post-graduate student, 5 post-graduate student, Novosibirsk state agrarian University
METHOD FOR THE STUDY OF NUCLEOTIDE SEQUENCES BLV
Abstract
For many years the study of BLV is an actual problem. So far not revealed many of the biological characteristics of the flow of the virus. We analyzed sequences of LTR-region 11 of BLV isolates, including circulating in livestock population on the territory of the Russian Federation. Picked up the most specific and sensitive primers flanking the most variable plot LTR-region.
Key words: bovine leukemia virus, genotyping, RFLP analysis, LTR-region, animal diseases, viruses, animal biotechnology
С каждым годом появляется все больше публикаций, посвящённых проблеме борьбы с лейкозом крупного рогатого скота. Некоторые авторы изучают эпизоотические особенности вируса, другие структурные. Однако проблема до настоящего времени остаётся более чем актуальной во всем мире [1-19].
Часто в оздоровленных ранее хозяйствах, племенных комплексах появляются новые вспышки инфекции. Данный факт интересует учёных во всем мире. Некоторые из них высказывают гипотезу о том, что вирус BLV находится в организме во временном «резервуаре», которым являются клетки крови, за исключением лимфоцитов [3-5, 9-11, 19]. Другие авторы утверждают, что на реализацию лейкозогенных потенций (тип течения) оказывают влияние структурные изменения BLV [1, 2, 4, 8, 14, 16-18]. Так, при совместных исследованиях группы ученых (Италии, Польши, Аргентины) были получены результаты, ученые сообщили, что на агрессивность вируса и тип течения инфекции оказывают влияние нуклеотидные замены в геноме вируса, которые находятся в CRE–области (являются транскрипционными активаторами GACGTCA, TGACG, GCA, TGACCTCA). Именно эти замены являются идентичными для BLV и HTLV провирусов и приводят к изменению поведения вируса, за исключением «TGACСTCA» замены в последовательности «TGACGTCA». Замена вышеуказанного нуклеотида в последовательности, на соответствующий в последовательности HTLV препятствует репликации вируса в естественных условиях [3, 15].
В трудах ряда учёных опубликованы последовательности олигонуклеотидных праймеров фланкирующих более обширный участок провируса BLV [6, 7, 12, 13] однако захватывающие, дополнительно ещё более 500 bp, что в итоге приводит к невосприимчивости праймеров (снижению чувствительности) к некоторым субтипам провируса, в связи, с чем возникла необходимость в конструировании более чувствительных праймеров.
Объектом исследования являлся крупный рогатый скот краснодарского края, Динского района. Материалом послужили пробы крови. Забор крови осуществляли при помощи стерильных вакуумных пробирок. В дальнейшем из крови выделяли ДНК, с использованием наборов AmpliSens согласно инструкции к наборам. Полимеразную цепную реакцию ставили в 25 мкл реакционной смеси: буфер (х10)- 5 мкл, смесь dNTP – 1 мкл, Pr1 – 1,5 мкл, Pr2 – 1,5 мкл, ДНК – 1 мкл, Taq-полимераза – 0,5 мкл, SG – 0,5 мкл., ТЕ-буфер до 25 мкл.
Результаты исследований
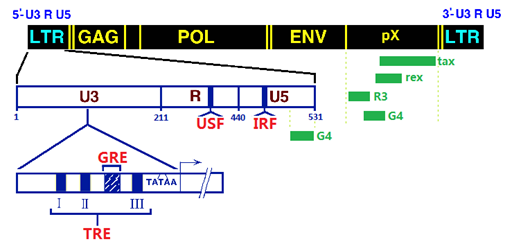
Мы проанализировали наиболее вариабельные участки типов и некоторых субтипов BLV, исключили их из областей фланкируемых праймерами. При помощи программы oligo (NBCI), подобрали последовательность праймеров 5' CAGAATTGGTTGCTAGCGGG 3'; 5'- CCA GAA GCG TTC TCC TCC TG -3', адаптировали по температурно-временным параметрам (рис. 1).
Рис. 1 – Схема участка провируса BLV фланкируемого олигонуклеотидными праймерами.
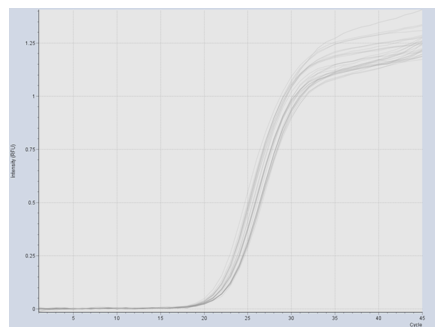
Поставили 3 реакции PCR с использованием красителя SuberGreen. После получения данных определили температуру плавления праймеров, которая составила 61 °C. Поставили контрольную реакцию с использованием профиля с рассчитанной температурой (рис 2).
Температурный профиль: 2 min – 95 °C; (30 s - 95 °C; 30 s - 61 °C; 30 s - 72 °C) – 40 циклов; 3 min – 72 °C.
Рис. 2 - График накопления копий провируса BLV с использованием праймеров, фланкирующих LTR-область (329 пн).
Далее опытным путём подбирали оптимальное соотношение концентрации праймеров к концентрации ДНК. Для этого поставили реакцию на четырех образцах в двух повторах с начальной концентрацией ДНК 50 нм, далее титровали в 10 раз. Визуально оценили, графики выхода продуктов амплификации. Поставили контрольную реакцию с рассчитанной температурой плавления и соотношением праймера к ДНК.
Данные олигонуклетидные праймеры можно использовать как для диагностики BLV, так и с целью получения участков провируса для дальнейшего изучения структурных особенностей методами ПДРФ анализа или секвинирования. Использование короткого участка провирусного генома является экономически более целесообразным методом изучения структурных особенностей BLV разных изолятов.
Литература
- Garcia-Etxebarria K., Jugo B.M. Evolutionary history of bovine endogenous retroviruses in the bovidae family BMC //Evolutionary Biology. 2013. Т. 13. № 1. С. 256.
- Ghysdael J, Bruck C, Kettmann R, Burny A (1984) Bovine Leukemia virus. Curr Top Microbiol Immunol 112: 1–19. doi: 10.1007/978-3-642-69677-0_1
- Gillet N, Florins A, Boxus M, Burteau C, Nigro A, et al. (2008) Mechanism of leukomogenesis induced by bovine leukemia virus: prospect for novel anti-retroviral therapies in human. Retrovirology 4: 18. doi: 10.1186/1742-4690-4-18
- Gutierrez G, Carignano H, Alvarez I, Martinez C, Porta N, Politzki R, Gammella M, Lomonaco M, Fondevila N, Poli M, Trono K: Bovine leukemia virus p24 antibodies reflect blood proviral load. BMC Vet Res 2012, 8:187.
- Jimba M, Takeshima SN, Murakami H, Kohara J, Kobayashi N, Matsuhashi T, Ohmori T, Nunoya T, Aida Y: BLV-CoCoMo-qPCR: a useful tool for evaluating bovine leukemia virus infection status. BMC Vet Res 2012, 8:167.
- Kamoi,K.,Yamamoto, K., Misawa, A., Miyake, A., Ishida, T., Tanaka, Y., Mochizuki, M., and Watanabe, T. (2006). SUV39H1 interacts with HTLV-1 Tax and abrogates Tax transactivation of HTLV-1 LTR. Retrovirology 13, 3-5.
- King AMQ, Adams J, Carstens EB, Lefkowitz EJ (2012) Virus Taxonomy: classification and nomenclature of viruses: Ninth Report of the ICTV. Amsterdam: Elsevier/Academic Press. 1344 p.
- Mansky LM, Temin HM (1994) Lower mutation rate of bovine leukemia virus relative to that of spleen necrosis virus. J Virol 68: 494–499.
- Meas S., Usui T., Ohashi K et al. Vertical transmission of bovine leukemia virus and bovine immunodeficiency virus in dairy cattle herds / Meas S., Usui T., Ohashi K., Sugimoto C., Onuma M. // Veterinary Microbiology . 2002. Т. 84. № 3. С. 275-282.
- Quantifying lymphocyte kinetics in vivo using carboxyfluorescein diacetate succinimidyl ester (CFSE) / Asquith B., Mosley A., Debacq C., Florins A., Gillet N., Sanchez-Alcaraz T., Willems L. // Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences. 2006. Т. 273.№ 1590. С. 1165-1171.
- Rates of CTL killing in persistent viral infection in vivo / Elemans M., Asquith B., Florins A., Willems L. // PLoS Computational Biology. 2014. Т. 10. № 4. С. e1003534.
- Rodríguez S.M. Preventive and therapeutic strategies for BLV: lessons for HTLV / Rodríguez S.M., Gillet N., Boulanger F., Willems L., Florins A., de Brogniez A., Sánchez-Alcaraz M.T., Boxus M., Gutiérrez G., Trono K., Alvarez I., Vagnoni L. // Viruses. 2011. Т. 3. № 7. С. 1210-1248.
- RiceNR, Simek SL, Dubois GC, Showalter SD,Gilden RV, Stephens RM: Expression of the bovine leukemia virus X region in virusinfected cells. J Virol 1987, 61:1577-1585.
- The molekular characterization of bovine leukemia virus isolates from eastern europe and siberia and its impact on phulogeny / Rola-Łuszczak M., Pluta A., Olech M., Kuźmak J., Donnik I., Petropavlovskiy M., Gerilovych A., Vinogradova I., Choudhury B. // PLoS ONE. 2013. Т. 8. № 3. С. e58705.
- Zhao X, McGirr KM, Buehring GC (2007) Evolutionary influences on overlapping reading frames in the bovine leukemia virus pXBL region. Genomics 89: 502–511. doi: 10.1016/j.ygeno.2006.12.007
- Батенёва Н.В., Смирнов П.Н., Михнович И.В. Изучение распространения условных генотипов вируса лейкоза крупного рогатого скота // Сельскохозяйственная биология. 2012, № 4, С.69-72
- Дробот Е.В.Результаты изучения генотипического разнообразия вируса лейкоза крупного рогатого скота и особенности эпизоотологического и гематологического проявления. Автореф. канд. дис. Новосибирск, 2007.
- Петропавловский М.В. Региональная молекулярно-генетическая структура вируса лейкоза крупного рогатого скота / Петропавловский М.В., Донник И.М. //Ветеринария Кубани. 2010. № 3. С. 12-13
- Смирнов П.Н.Болезнь века -лейкоз крупного рогатого скота.-Новосибирск, 2007.
References
- Garcia-Etxebarria K., Jugo B.M. Evolutionary history of bovine endogenous retroviruses in the bovidae family BMC //Evolutionary Biology. 2013. Т. 13. № 1. С. 256.
- Ghysdael J, Bruck C, Kettmann R, Burny A (1984) Bovine Leukemia virus. Curr Top Microbiol Immunol 112: 1–19. doi: 10.1007/978-3-642-69677-0_1
- Gillet N, Florins A, Boxus M, Burteau C, Nigro A, et al. (2008) Mechanism of leukomogenesis induced by bovine leukemia virus: prospect for novel anti-retroviral therapies in human. Retrovirology 4: 18. doi: 10.1186/1742-4690-4-18
- Gutierrez G, Carignano H, Alvarez I, Martinez C, Porta N, Politzki R, Gammella M, Lomonaco M, Fondevila N, Poli M, Trono K: Bovine leukemia virus p24 antibodies reflect blood proviral load. BMC Vet Res 2012, 8:187.
- Jimba M, Takeshima SN, Murakami H, Kohara J, Kobayashi N, Matsuhashi T, Ohmori T, Nunoya T, Aida Y: BLV-CoCoMo-qPCR: a useful tool for evaluating bovine leukemia virus infection status. BMC Vet Res 2012, 8:167.
- Kamoi,K.,Yamamoto, K., Misawa, A., Miyake, A., Ishida, T., Tanaka, Y., Mochizuki, M., and Watanabe, T. (2006). SUV39H1 interacts with HTLV-1 Tax and abrogates Tax transactivation of HTLV-1 LTR. Retrovirology 13, 3-5.
- King AMQ, Adams J, Carstens EB, Lefkowitz EJ (2012) Virus Taxonomy: classification and nomenclature of viruses: Ninth Report of the ICTV. Amsterdam: Elsevier/Academic Press. 1344 p.
- Mansky LM, Temin HM (1994) Lower mutation rate of bovine leukemia virus relative to that of spleen necrosis virus. J Virol 68: 494–499.
- Meas S., Usui T., Ohashi K et al. Vertical transmission of bovine leukemia virus and bovine immunodeficiency virus in dairy cattle herds / Meas S., Usui T., Ohashi K., Sugimoto C., Onuma M. // Veterinary Microbiology . 2002. Т. 84. № 3. С. 275-282.
- Quantifying lymphocyte kinetics in vivo using carboxyfluorescein diacetate succinimidyl ester (CFSE) / Asquith B., Mosley A., Debacq C., Florins A., Gillet N., Sanchez-Alcaraz T., Willems L. // Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences. 2006. Т. 273.№ 1590. С. 1165-1171.
- Rates of CTL killing in persistent viral infection in vivo / Elemans M., Asquith B., Florins A., Willems L. // PLoS Computational Biology. 2014. Т. 10. № 4. С. e1003534.
- Rodríguez S.M. Preventive and therapeutic strategies for BLV: lessons for HTLV / Rodríguez S.M., Gillet N., Boulanger F., Willems L., Florins A., de Brogniez A., Sánchez-Alcaraz M.T., Boxus M., Gutiérrez G., Trono K., Alvarez I., Vagnoni L. // Viruses. 2011. Т. 3. № 7. С. 1210-1248.
- RiceNR, Simek SL, Dubois GC, Showalter SD,Gilden RV, Stephens RM: Expression of the bovine leukemia virus X region in virusinfected cells. J Virol 1987, 61:1577-1585.
- The molekular characterization of bovine leukemia virus isolates from eastern europe and siberia and its impact on phulogeny / Rola-Łuszczak M., Pluta A., Olech M., Kuźmak J., Donnik I., Petropavlovskiy M., Gerilovych A., Vinogradova I., Choudhury B. // PLoS ONE. 2013. Т. 8. № 3. С. e58705.
- Zhao X, McGirr KM, Buehring GC (2007) Evolutionary influences on overlapping reading frames in the bovine leukemia virus pXBL region. Genomics 89: 502–511. doi: 10.1016/j.ygeno.2006.12.007
- Bateneva N. V., Smirnov P. N., Michnovicz I. V. study of the conditional distribution of genotypes of the virus leukemia cattle / Agricultural biology. 2012, No. 4, Pp. 69-7
- Drobot, E. V. The results of the study of genotypic diversity of leukemia virus of cattle and epizootological features and hematologic manifestations. Author. Cand. dis. Novosibirsk, 2007.
- Peteropavlovskiy M. V. Regional molecular genetic structure of the virus leukemia cattle / Peteropavlovskiy M. V., Donnik I. M. //veterinary Kuban. 2010. No. 3. S. 12-13
- Smirnov P. N. The disease of the century-the bovine leukemia.-Novosibirsk, 2007.