АНАЛИЗ СТРУКТУРНОГО РАЗНООБРАЗИЯ ПРОВИРУСА BLV НА ОСНОВАНИИ ТЕМПЕРАТУРНЫХ КРИВЫХ REAL TIME SYBRGREEN
Смирнов П.Н.
Доктор ветеринарных наук, профессор, Новосибирский государственный аграрный университет
Батенёва Н.В.
Кандидат биологических наук, Новосибирский государственный аграрный университет
АНАЛИЗ СТРУКТУРНОГО РАЗНООБРАЗИЯ ПРОВИРУСА BLV НА ОСНОВАНИИ ТЕМПЕРАТУРНЫХ КРИВЫХ REAL TIME SYBRGREEN
Аннотация
Исследования направлены на изучение структурного разнообразия провируса BLV на основании температурных кривых Real Time SYBRgreen. По нуклеотидному составу, на основании температурных пиков, выборки распределились на несколько групп, соответствующих международной классификации генотипов провируса BLV (I-VII).
Ключевые слова: вирус лейкоза крупного рогатого скота, генотипирование, анализ температурных кривых, олигонуклеотидные праймеры, анализ структурного разнообразия.
Smirnov P.N.
Doctor of Veterinary Sciences, Professor, Novosibirsk State Agrarian University
Bateneva N.V.
Candidate of Biological Sciences, Novosibirsk State Agrarian University
ANALYSIS OF THE STRUCTURAL DIVERSITY OF PROVIRUS BLV ON THE BASIS OF TEMPERATURE CURVES REAL TIME (SYBRGREEN)
Abstract
Researches Investigations are directed on studying of the structural diversity of provirus BLV on the basis of the temperature curve in real time SYBRgreen. Studied blood samples were collected from cattle farms of the Krasnodar region. On the above mentioned farms contained cows the following breeds: Red Steppe, Ireshire, Holstaine. The blood sample was divided into several groups according to the nucleotides structure, based on temperature peaks, who match international classification of provirus’s genotypes BLV (I-VII). Knowing that peaks depending on the temperature at different genotypes provirus’s BLV, possibly, at genotyping not use restriction fragment length polymorphism (RFLP) or sequencing.
Keywords: BLV (bovine leukemia virus), leucosis, genotypes, provirus BLV, temperature curves, analysis of the structural diversity, cattle, oligonucleotide primers.
В организме хозяина хронически персистирующий вирус может претерпевать мутационные изменения, направленные на увеличение патогенности, на преодоление более эффективных защитных механизмов хозяина. Поэтому, наряду с генетическим статусом макроорганизма, во внимание необходимо принимать и генетическую изменчивость самого патогена, который, возможно, в процессе длительной персистенции способен изменять свою патогенность, в сторону как комменсализма, так и усиления патогенных свойств [2, 3, 5]. Генетическая вариабельность BLV у инфицированного крупного рогатого скота достаточно хорошо охарактеризована [1, 4, 6, 8]. На основании метода ПДРФ и ДНК секвенирования вируса лейкоза крупного рогатого скота классифицировано три подгруппы вируса — бельгийская, австралийская и японская [4, 6, 10]. Так как секвинирование достаточно затратный метод генотипирования, для распределения выборки по известным генотипам применяют ПДРФ-анализ (полиморфизм длин ристрикционных фрагментов) или методику Real time PCR с флуоресцентно-мечеными олигонуклеотидными зондами. Наиболее широко используется 5'-экзонуклеазный метод, основанный на эффекте пространственного разделения флуорофора и гасителя флуоресценции вследствие расщепления Taq ДНК-полимеразой зонда, связанного с амплифицируемой последовательностью ДНК [7].
Учитывая вышесказанное целью наших исследований явилось проведение генотипирования BLV-провируса по env гену циркулирующего на поголовье крупного рогатого скота Краснодарского края, с распределением исследуемых образцов по структурной составляющей на группы по gag гену. В связи с этим были поставлены следующие задачи: 1) выделить ДНК из крови серопозитивных животных; 2) провести ПЦР в реальном времени; построить графики температур плавления и накопления продуктов амплификации; 3) провести биоинформационный анализ полученных данных.
Объектом исследований являлся крупный рогатый скот принадлежащий ПЗ «им.Чапаева», Краснодарского края. Материалом послужила кровь серопозитивных животных в отношение BLV (bovine leukemia virus). ДНК из крови выделяли сорбентным методом с использованием наборов МедиГен (Новосибирск).
Для определения вирусоносителя по гену env gp51 применили следующие праймеры:№ 1. 5´TCTGTGCCAAGTCTCCCAGATA-3´, № 2. 5´СССACAAGGGCGGCGCCGGTTT-3´, [2, 3]; для определения вирусоносителя по гену gag p24 применили следующие праймеры (347 bp): № 1. 5’GGAGGWGGRAAGATGCGAACTATT 3’, № 2. 5’GTCCGYTCTA CYAACCCTGAACTT 3’ [9], синтезированные ООО «Меди-ген». Применяли способ проведения PCR real time с SYBRgreen-II.
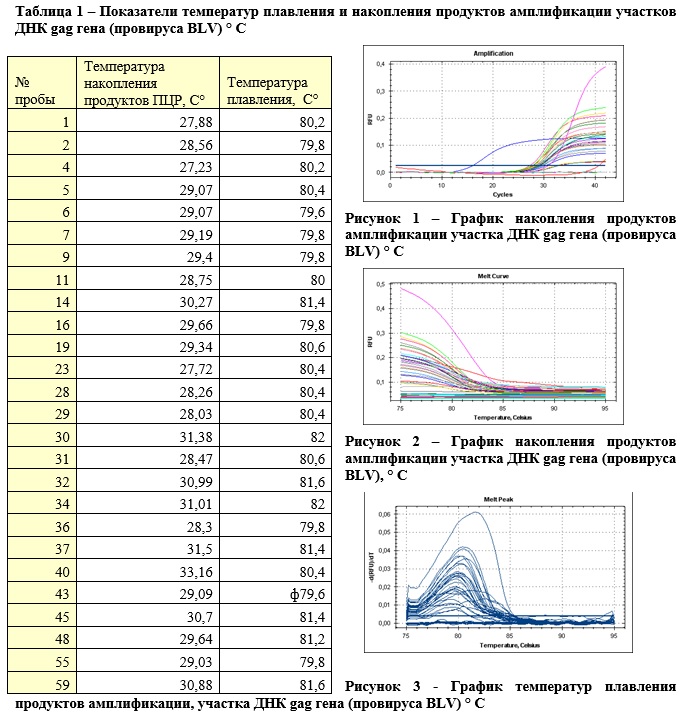
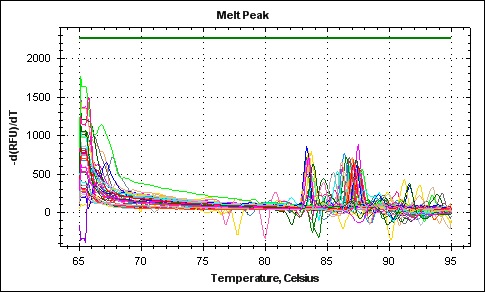
На рисунке 1 изображен график ПЦР real-time, в котором отображается накопление специфических продуктов амплификации BLV. При этом максимальное количество продуктов амплификации было отмечено при температуре 28 до 32º С. Однако, температурная кривая некоторых проб значительно отличалась. На следующем графике (рис. 2) изображены кривые температур плавления амликонов, а на рисунке 3 изображен участок кривых, где отмечалась наибольшая изменчивость (t 75-95 º С). Стоит отметить, что для данного аплифицируемого участка вышеуказанной выборки оптимальная температура плавления составляет от 79,6 до 81,4ºС. Определив опытным путем оптимальные температуры плавления и накопления продуктов амплификации, были адаптированы режимы аплификации, что позволит максимально исключить ложноположительные результаты.
Впоследствии был проведен более точный анализ - НРМ (по кривым плавления), результаты которого напрямую зависят от нуклеотидного состава, а, соответственно, и генотипов исследуемых образцов.
Конкретный профиль плавления, который отражает кривые плавления, определяется последовательностью оснований в исследуемой ДНК. Пики на дифференциальных кривых плавления связаны с выплавлением участков молекулы в интервале в несколько десятых градуса отдельных с характерным размером в несколько сотен пар оснований
Рисунок 4 – График температур плавления продуктов амплификации env гена (провируса BLV), ° С
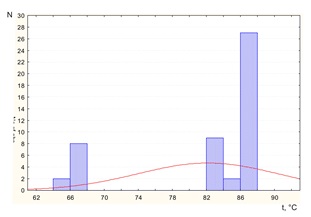
Расхождение пиков температур плавления не принципиальны (Табл. 2, Рис. 4), а, соответственно, данные образцы можно отнести к одному генотипу, в то время как у этих же животных наблюдались значительные нуклеотидные замены на исследуемом участке ДНК провируса другого гена (gag) (Табл. 1, Рис. 1-3). Данный факт лишь подтверждает гипотезу о несоответствии генотипической принадлежности изучаемого провируса по двум генам. Однако четко видны повторяющиеся на каждом из образцов температурные пики, что свидетельствует о распределении выборки на несколько групп по нуклеотидному составу. На графике четко видны пять групп, с температурами плавления от 64 до 88 °С. Так к первой группе отнесли образцы, температура плавления которых составила 64-66, ко второй – 66,1-68, к третей – 82-84, к четвертой – 84,1-86 и к пятой -86, 1-88 °С. Самой многочисленной оказалась группа 5, далее по убыванию третья, вторая, первая и четвертая. Проведя биоинформационный анализ данных образцов мы определили, что данные группы соответствуют международной классификации генотипов (I-VII) [6, 8].
Рисунок 5 – Распределение исследуемых образцов на группы по структуре ДНК
(участок 444 п.н. env ген)
Рисунок 6 – Распределение исследуемых образцов на группы по структуре ДНК
(участок 347 п.н. gag ген)
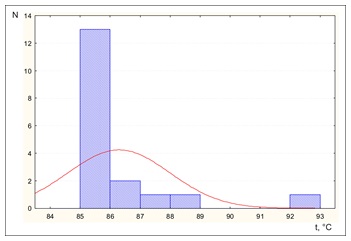
Анализируя полученные данные по температурным кривым образцов исследуемых по gag гену BLV (провируса), следует отметить, что образцы также распределились на 5 групп, однако совершенно в других температурных рамках. Так зарегистрировано 5 групп: первая 85-86, вторая – 86, 1-87, третья – 87,1-88, четвертая 88,1-89, пятая 92-93 °С. Характеризуя данные группы, напрашивается вывод об определенной гомогенности 1-4 групп, и совершенно отличающейся температурами плавления образцов, входящих в 5 группу. Данный факт указывает на большое содержание в структуре ДНК, С (цитозин) и G (гуанин), которые способны образовывать тройные связи, а соответственно при их плавлении необходимы более высокие температуры. Так как на данный момент не существует определенной классификации генотипов, по данному участку gag гена, в дальнейшем предстоит провести секвенирование нескольких образцов из каждой группы.
Выводы и перспективы дальнейших исследований.
- Биоинформационным анализом результатов исследований ДНК провирусов BLV от крупного рогатого скота Краснодарского края выявлено 5 условных генотипов.
- Распределение выборки, по температуре плавления, неравномерное. Наиболее многочисленной оказалась группа – 86, 1-880; далее в хронологическом порядке – 82 -840; 66, 1-68; 64 – 880 и 84,1 – 860 по gag гену.
Список литературы
Грачева Н.В. Генотипическое разнообразие вируса лейкоза крупного рогатого скота по gag и env генам: автореф. дис…. канд. биол. наук./ Н.В. Грачева. – Новосибирск, 2010. – 18 с.
Дробот, Е.В. Результаты изучения генотипического разнообразия вируса лейкоза крупного рогатого скота и особенности эпизоотологического и гематологического проявления: автореф. дис…. канд. биол. наук./ Е.В. Дробот. – Новосибирск, 2007. – 18 с.
Смирнов, П.Н. Болезнь века – лейкоз крупного рогатого скота. – 2007. – 301 с.
Beyer, J.R., R.B. Kollner and J.P. Teifke, 2002. Cattle infected with bovine leukaemia virus may not only develop persistent B-cell lymphocytosis but also persistent B-cell lymphopenia. Journal of Veterinary, 49(6): 270-276
Burny, A., 1988. Bovine leukemia: fact and hypotheses derived from the study of an infections cancer. Veterinary Microbiology, 17: 197-218.
Fecher, H., A. Kurg and L. Geue, 2003. Evaluation of polymerase chain reaction (PCR) application in diagnosis of bovine leukemia virus (BLV) infection in naturally infected cattle. Journal of American Veterinary Medical Association, 222(7): 983-985.
Heid, S.A., J. Stevens, К.J. Livak, and Р.М. Williams. 1996. Real time quantitative PCR. Genome Res. 6: 986-994
Licursi, M., Y. Inoshima, T. Yokoyama and E. Gonzales, 2002. Genetic heterogeneity bovine leukemia virus genotypes and its relation to humoral responses in hosts. Virus Research, 86: 101-110.
Mohammadabadi M.R., G.O. Shaikaev and G.E. Sulimova, 2004. Detection of bovine leukemia virus proviral DNA in Yaroslavsl, Mongolian and black pied cattle by PCA. Cellular & Molecular Biology letters, 9 (4): 766-768.
Monti, G., R. Schrijver and D. Bier, 2005. Genetic diversity and spread of Bovine leukaemia virus isolates in Argentine dairy cattle. Arch Virol, 150 (3): 443-458.