ИССЛЕДОВАНИЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ D-ПЕТЛИ МТДНК У ОВЕЦ МЕРИНОСОВЫХ ПОРОД РФ
ИССЛЕДОВАНИЕ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ D-ПЕТЛИ МТДНК У ОВЕЦ МЕРИНОСОВЫХ ПОРОД РФ
Научная статья
Широкова Н.В.1, *, Бакоев Н.Ф.2, Колосова М.А.3, Гетманцева Л.В.4 , Колосов Ю.А.5
1 ORCID: 0000-0002-5607-3044;
2 ORCID: 0000-0001-5495-8191;
3 ORCID: 0000-0003-2979-7108;
4 ORCID: 0000-0003-1868-3148;
5 ORCID: 0000-0002-6826-8009;
1, 3, 5 Донской государственный аграрный университет, Персиановский, Россия;
2, 4 Всероссийский институт животноводства им. Л.К. Эрнста, Подольск, Россия
* Корреспондирующий автор (m.leonovaa[at]mail.ru)
Аннотация
На сегодняшний день мериносовые породы разводят на различных континентах, однако в процессе породообразования, с учетом климатических и экономических особенностей, породы приобрели свои уникальные особенности. Целью данной работы стало изучение нуклеотидной последовательности D-петли мтДНК у овец мериносовых пород Южного региона РФ и поиск вариативных локусов, обуславливающих сходство и различия между мериносовыми породами отечественной и зарубежной селекции. В результате исследований установлено 20 вариабельных нуклеотидных позиций участка D-петли мтДНК, обуславливающих сходство и различия этих пород с мериносами европейской и австралийской селекции.
Ключевые слова: мтДНК, D-петля, вариабельность, мериносовые породы, овцы.
STUDY OF NUCLEOTIDE SEQUENCE OF MTDNA D-LOOP IN MERINO SHEEP BREEDS OF THE RUSSIAN FEDERATION
Research article
Shirokova N.V.1, *, Bakoev N.F.2, Kolosova M.A.3, Getmantseva L.V.4,Kolosov Yu.A.5
1 ORCID: 0000-0002-5607-3044;
2 ORCID: 0000-0001-5495-8191;
3 ORCID: 0000-0003-2979-7108;
4 ORCID: 0000-0003-1868-3148;
5 ORCID: 0000-0002-6826-8009;
1, 3, 5 Don State Agrarian University, Persianovsky, Russia;
2, 4 L.K. Ernst Institute of Animal Husbandry, Podolsk, Russia
* Corresponding author (m.leonovaa[at]mail.ru)
Abstract
Merino breeds are reared on different continents, but each breed acquired its own unique features in the process of breed formation with regard to climatic and economic features. The aim of this work was to study the nucleotide sequence of the mtDNA D-loop in sheep of Merino breeds of the Southern region of the Russian Federation and to search for variable loci, which cause similarities and differences between the Merino breeds of domestic and foreign selection. As a result of the research, 20 variable nucleotide positions of the mtDNA D-loop region, determining the similarities and differences of these breeds of merino sheep of European and Australian rearing, are established.
Keywords: mtDNA, D-loop, variability, merino breeds, sheep.
Развитие мирового овцеводства подвержено влиянию промышленности, процессов интенсификации сельского хозяйства и других непрерывных изменений, сопровождающих наше общество. Большой спрос на высококачественную тонкую шерсть способствовали появлению овец мериносовых и тонкорунных пород. Прародителями мериносовых пород являются овцы, разводившиеся в древности в Малой Азии, которые со временем получили распространение в Средиземноморской части Европы. С середины IV века и до конца XVI века Испания была мировым монополистом по мериносовому овцеводству. С прекращением испанской монополии тонкорунное овцеводство получило свое развитие в европейских странах, а в дальнейшем и по всем миру [1], [2], [3].
В Россию мериносовые овцы были завезены в XVIII - XIX столетиях из европейских стран. Это были очень нежные животные, нуждающиеся в особых условиях кормления и содержания, и русские овцеводы начали создавать породы овец, более приспособленные к местным условиям. Породы сальская, ставропольская и советский меринос были созданы в период 1920-1950 гг. путем скрещивание мазаевских и новокавказских маток с баранами породы рамбулье и дальнейшее их совершенствование проводилось в соответствии с заданными параметрами, обуславливающими межпородные различия [1], [6].
Создание породы австралийского мериноса также происходило на основе европейских мериносов, после окончания Испанской монополии. Однако, согласно работе Fan H. Et al. [4], первые овцы, попавшие в Австралию, были с мыса Доброй Надежды. Они были белого цвета с относительно широкими длинными хвостами [5]. В начале 1800-х годов в Австралию стали завозить европейских мериносов [7]. Поначалу особый упор был все-таки на получения мясной продукции, что давало преимущества кроссированным животным, отличающимся массой и плодовитостью. В дальнейшем уже селекционные работы были направлены на получение высококачественной шерсти, которая пользовалась очень большим спросом [8], [9], [10].
Все эти особенности происхождения могли определенным образом отразиться на генетической конструкции мтДНК мериносовых пород австралийской селекции и обусловить различия, относительно мериносовых пород европейской селекции.
Цель
Целью нашего исследования стало изучение нуклеотидной последовательности участка D-петли мтДНК у овец мериносовых пород Южного региона РФ (сальской, ставропольской и советский меринос) и поиск вариативных локусов, обуславливающих сходство и различия между мериносовыми породами отечественной и зарубежной селекции.
Материалы и методы
Исследование мтДНК проводили на овцах породы сальская (n=11), ставропольская (n=8) и советский меринос (n=12). МтДНК выделяли из образцов ткани с использованием набора «К-Сорб-100» (ООО «Синтол») в соответствии с инструкцией производителя. ПЦР проводили по стандартной методике [4]. Для амплификации фрагментов D-петли мтДНК длиной 1352 п.н. использовали праймеры: F 5’- GGT CTT GTA AAC CAG AGA AGG AG - 3’ и R 3’- TGG AGT CAG TAG ACT CAT CTA GG - 5’. Услуги по секвенированию фрагментов оказывала компания «Евроген». Редактирование и выравнивание последовательностей проводили при помощи программ BioEdit v7.2.6 и MEGA 7. Референсной была выбрана последовательность Accession NC_001941.1 NCBI.
Для проведения сравнительного анализа из базы NCBI (англ. National Center for Biotechnological Information), были взяты последовательности участка D-петли мтДНК, принадлежащие породам овец: австралийский меринос (HM236174.1) и мериноландшаф (NC_001941.1). Для определения принадлежности исследуемых образцов к гаплогруппам были выбраны последовательности D-петли мтДНК, относящиеся к гаплогруппам А (DQ852286), В (DQ852282), С (DQ852284), D (DQ852288) и Е (DQ852280).
Результаты исследований и их обсуждение
В результате исследований были получены фрагменты мтДНК области D-петли длиной 1179 п.н. и определена первичная структура нуклеотидов между позициями 15437-16616 п.н. у овец пород сальская, ставропольская и советский меринос. У всех исследуемых животных были установлены 4 тандемных повтора длиной 75 п.н. Построение филогенетического дерева методом максимального правдоподия показало четкое разделение исследуемых овец на два кластера. При этом первый кластер относился к гаплогруппе В, характерной для европейских домашних овец, а второй кластер к гаплогруппе А, доминирующей у азиатских овец, а также Новой Зеландии и Австралии (рис.1).
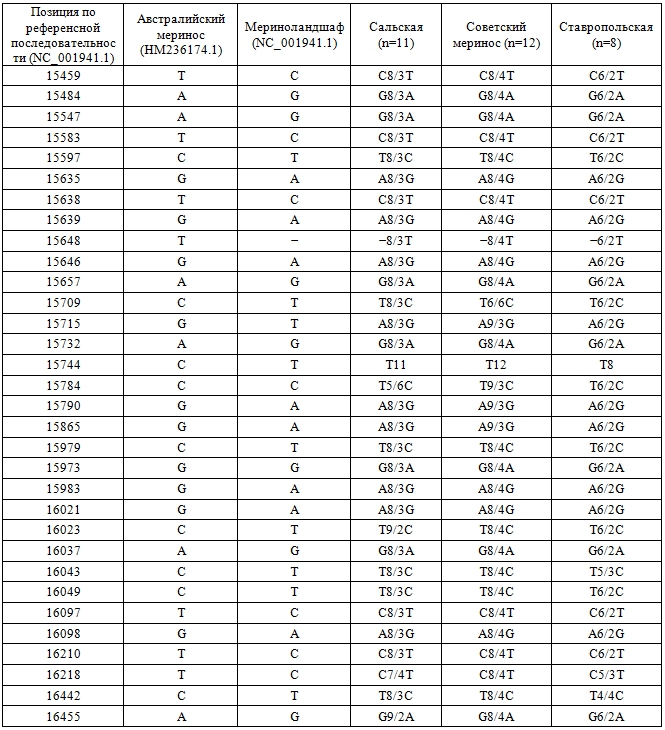
В целом, относительно последовательности австралийского мериноса у исследуемых овец были определены 32 вариабельные нуклеотидные позиции, из них 20 позиций были четко разделены на два гаплотипа во всех трех породах. Первый гаплотип был идентичен с породой мериноландшаф (европейские мериносы), а второй с австралийским мериносом (табл.). Относительно других позиций следует отметить, что у всех исследуемых овец в позиции 15744 п.н. был определен нуклеотид Т. Вариабельность остальных сайтов можно расматривать как внутрипородный полиморфизм.

Рис. 1 – Филогенетические отношения между исследуемыми овцами пород сальская, ставропольская и советский меринос и гаплогруппами А, В, С, D, E построены с использованием метода максимального правдоподобия, основанного на модели Тамура-Ней
Полученные нами результаты показывают, что в нашей выборке овец мериносовых пород, разводимых в Южном регионе РФ (сальской, ставропольской и советский меринос) фрагмент мтДНК D-петли в большей степени идентичен европейским мериносам. Однако, в среднем у 30% исследуемых овец прослеживается гаплотип, характерный для мериносовых овец австралийской селекции.
Таблица 1 – Различия нуклеотидной последовательности D-петли мтДНК у мериносовых пород отечественной и зарубежной селекции
Следует отметить, что использование баранов австралийской селекции для совершенствования мериносовых пород РФ не отражается на генетической структуре мтДНК исходного поголовья. Изменения в мтДНК, а точнее закрепление мтДНК, свойственной мериносам австралийской селекции, у мериносовых пород РФ, может произойти только в случае использования овцематок австралийского мериноса и дальнейшего закрепления таких дочерей в популяции.
Выводы
Таким образом, изучение нуклеотидной последовательности D-петли мтДНК у овец мериносовых пород Южного региона РФ (сальской, ставропольской и советский меринос) позволило выявить 20 вариабельных нуклеотидных позиций участка D-петли мтДНК. Построение филогенетического дерева методом максимального правдоподия показало разделение исследуемых овец на два кластера. При этом первый кластер представлен гаплогруппой В, характерной для европейских домашних овец, а второй кластер относится к гаплогруппе А, доминирующей у азиатских овец, а также Новой Зеландии и Австралии. Полученные результаты исследований позволили установить сходство и различие овец мериносовых пород Южного региона РФ с мериносами европейской и австралийской селекции.
| Финансирование Работа выполнена при поддержке гранта Президента Российской Федерации №14.W01.17.1030-MK. | Funding This work was supported by the grant of the President of the Russian Federation No. 14.W01.17.1030-MK. |
| Конфликт интересов Не указан. | Conflict of Interest None declared. |
Список литературы / References
- Колосов Ю. А. Использование генофонда мериносовых овец отечественной и импортной селекции для совершенствования местных мериносов. Овцы, козы, шерстное дело / Ю. А. Колосов .- 2012.- №4.- С.13-16.
- Широкова Н. В. Полиморфизм микросателлитных локусов OARCP549, CSRD247, FCB20 И MAF65 у овец. / Н.В. Широкова, Л. В. Гетманцева, Ю .А. Колосов // Аграрная наука Евро-Северо-Востока. 2017. № 5 (60). С. 57-62.
- Chen X. Mitochondrial DNA T7719G in tRNA-Lys gene affects litter size in Small-tailed Han sheep / X. Chen, D. Wang, H. Xiang and others // Journal of Animal Science and Biotechnology 2017; 8:31.
- Fan H. Complete Mitochondrial Genome Sequences of Chinese Indigenous Sheep with Different Tail Types and an Analysis of Phylogenetic Evolution in Domestic Sheep / H. Fan, F. Zhao, C. Zhu and others // Asian Australas. J. Anim. Sci. 2016; 29 (5) P. 631-639.
- Gorkhali N. A. Mitochondrial DNA Variation in Indigenous Sheep (Ovis aries) Breeds of Nepal Tropical / N. A. Gorkhali, J. L. Han, Y. H. Ma // Agricultural Research; 2015; 26 (4) P. 632-641.
- Gorlov I. F. CAST/MspI gene polymorphism and its impact on growth traits of soviet merino and salsk sheep breeds in the south european part of Russia / I. F. Gorlov, N. V. Shirokova, A. V. Randelin // Turkish Journal of Veterinary and Animal Sciences. 2016. Т. 40. № 4. P. 399-405.
- Gorlov I. F. Association of the growth hormone gene polymorphism with growth traits in salsk sheep breed / I. F. Gorlov, N. V. Shirokova, M. I. Slozhenkina and others // Small Ruminant Research. 2017. Т. 150. P. 11-14.
- Meadows J. R. S. Haplogroup relationships between domestic and wild sheep resolved using a mitogenome panel. Heredity / J. R. S. Meadows, S. Hiendleder, J. W. Kijas, 2011, 106 P. 700-706.
- Tapio M. Sheep mitochondrial DNA variation in European, Caucasian, and Central Asian areas / M. Tapi, N. Marzanov, M. Ozerov and others. 2006.. Mol Biol Evol. 2006, . 23. P. 1776–1783.
- Yilmaz O. Polymorphism of the ovine calpastatin gene in some Turkish sheep breeds / O. Yilmaz, T. Sezenler, N. Ata and others // Turk J Vet Anim Sci. 2014, 38 P. 354-357.
Список литературы на английском языке / References in English
- Kolosov Yu. A. Ispol'zovanie genofonda merinosovyh ovec otechestvennoj i importnoj selekcii dlya sovershenstvovaniya mestnyh merinosov. Ovcy, kozy, sherstnoe delo [Using the gene pool of merino sheep domestic and imported breeding to improve the local merino. Sheep, goats, wool business] /Yu. A. Kolosov.- 2012.- №4.- P.13-16.
- Shirokova N. V. Polimorfizm mikrosatellitnyh lokusov OARCP549, CSRD247, FCB20 I MAF65 u ovec [Polymorphism of microsatellite loci OarCP549, CSRD247, FCB20 and MAF65 in sheep] / N. V. Shirokova, L. V. Getmantseva,Yu. A. Kolosov and others // Agrarnaya nauka Evro-Severo-Vostoka [Agrarian Science of Euro-Northeast]. 2017. No. 5 (60). P. 57-62.
- Chen X. Mitochondrial DNA T7719G in tRNA-Lys gene affects litter size in Small-tailed Han sheep / X. Chen, D. Wang, H. Xiang and others // Journal of Animal Science and Biotechnology 2017; 8:31.
- Fan H. Complete Mitochondrial Genome Sequences of Chinese Indigenous Sheep with Different Tail Types and an Analysis of Phylogenetic Evolution in Domestic Sheep / H. Fan, F. Zhao, C. Zhu and others // Asian Australas. J. Anim. Sci. 2016; 29 (5) P. 631-639.
- Gorkhali N. A. Mitochondrial DNA Variation in Indigenous Sheep (Ovis aries) Breeds of Nepal Tropical / N. A. Gorkhali, J. L. Han, Y. H. Ma // Agricultural Research; 2015; 26 (4) P. 632-641.
- Gorlov I. F. CAST/MspI gene polymorphism and its impact on growth traits of soviet merino and salsk sheep breeds in the south european part of Russia / I. F. Gorlov, N. V. Shirokova, A. V. Randelin // Turkish Journal of Veterinary and Animal Sciences. 2016. Т. 40. № 4. P. 399-405.
- Gorlov I. F. Association of the growth hormone gene polymorphism with growth traits in salsk sheep breed / I. F. Gorlov, N. V. Shirokova, M. I. Slozhenkina and others // Small Ruminant Research. 2017. Т. 150. P. 11-14.
- Meadows J. R. S. Haplogroup relationships between domestic and wild sheep resolved using a mitogenome panel. Heredity / J. R. S. Meadows, S. Hiendleder, J. W. Kijas, 2011, 106 P. 700-706.
- Tapio M. Sheep mitochondrial DNA variation in European, Caucasian, and Central Asian areas / M. Tapi, N. Marzanov, M. Ozerov and others. 2006.. Mol Biol Evol. 2006, . 23. P. 1776–1783.
- Yilmaz O. Polymorphism of the ovine calpastatin gene in some Turkish sheep breeds / O. Yilmaz, T. Sezenler, N. Ata and others // Turk J Vet Anim Sci. 2014, 38 P. 354-357.